¡Precaución!
No utilice Internet Explorer para este laboratorio, ya que OpenRefine no funciona bien en este navegador.
Objetivo
Utilizar OpenRefine para administrar, validar y limpiar de manera eficiente datos sobre biodiversidad, asegurando una mejor calidad para su publicación.
Introducción
Sobre la Herramienta
OpenRefine es un software creado con el objetivo de pulir datos crudos hasta convertirlos en diamantes, los cuales son activos valiosos en la era del BigData. Este programa permite visualizar y manipular datos tabulares, facilitando el mejoramiento de la calidad general de un conjunto de datos. Tiene la apariencia de un software tradicional de hoja de cálculo (similar a Excel), pero funciona como una base de datos. Esto significa que OpenRefine no es adecuado para adicionar nuevas filas de datos, pero es extremadamente poderoso cuando se trata de explorar, limpiar y vincular datos.
OpenRefine es un software de código abierto que trabaja bajo una licencia BSD y se instala localmente, por lo que funciona como una aplicación web personal y de acceso privado a la que se accede desde un navegador web. Esta herramienta sirve para todo tipo de datos. Sin embargo, en este laboratorio se explica su funcionamiento en el contexto de datos sobre biodiversidad, estandarizados en Darwin Core. Puede consultar más información de la herramienta en el manual de usuario (en inglés).
Convenciones
Requerimientos
Archivo de trabajo
Asegúrese de que su navegador predeterminado sea Google Chrome o Mozilla Firefox.
Puede cargar datos con diferentes formatos y extensiones: TSV, CSV, SV, Excel (.xls y .xlsx), JSON, XML, RDF as XML y datos de Google Docs.
Abra OpenRefine y seleccione la pestaña Create Project. Para cargar el archivo, siga la ruta Get data from > This Computer y haga clic en Choose Files (Fig. 1).
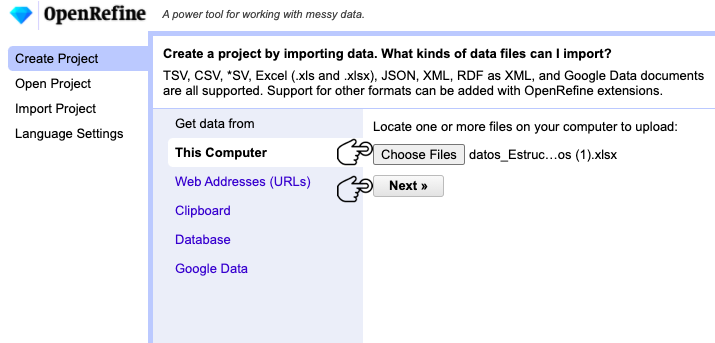 Figura 1. Creación de un proyecto en OpenRefine.
Figura 1. Creación de un proyecto en OpenRefine.
Seleccione el archivo «datos_Estructurados.xlsx» que descargó al incio del laboratorio y haga clic en Next (Fig. 1).
Puede subir varios archivos a la vez, el software unirá los archivos con base en los encabezados de las columnas.
Tras seleccionar el archivo, aparecerá un panel de configuración. Este permite especificar el tipo de archivo que cargó y configurar la manera en la que los datos son leídos (Fig. 2).
OpenRefine hace una interpretación automática del tipo de archivo, la codificación del texto, las filas de encabezado, entre otros. El conjunto de datos de ejemplo de este laboratorio es interpretado de manera correcta y no necesita ajustes adicionales.
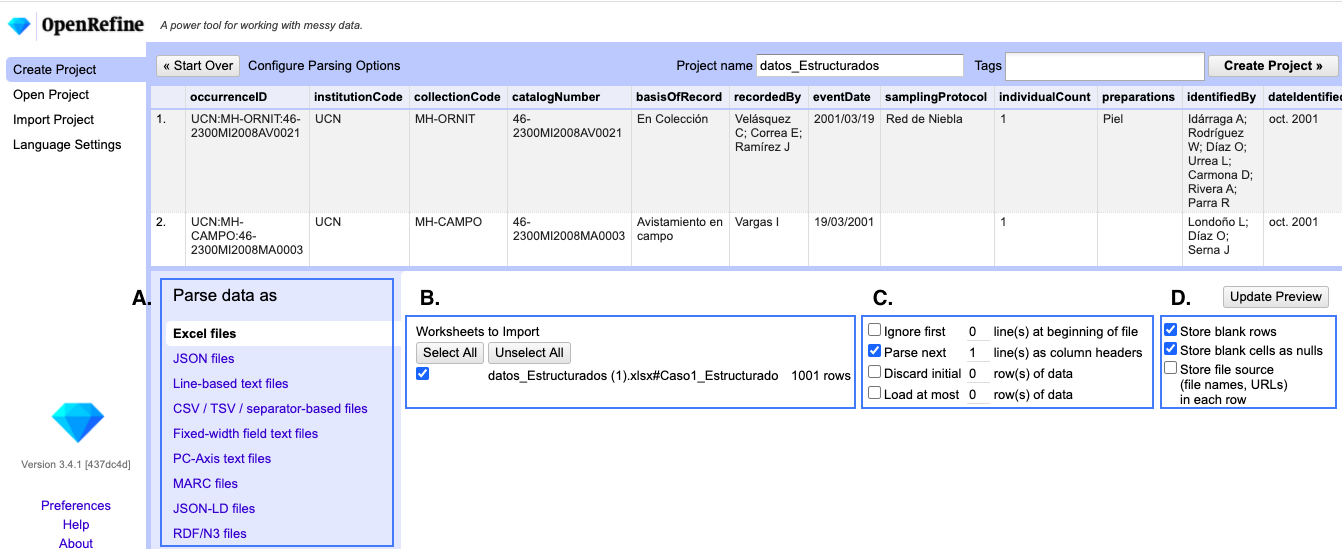
Figura 2. Opciones para configurar la lectura y carga de los datos para un archivo Excel (.xls y .xlsx): A. Tipo de archivo, B. Hojas a importar, C. Elección de filas, D. Carga de filas.
Character encoding: al cargar datos en formato de texto plano, es posible que la previsualización muestre las tíldes (í) y las eñes (ñ) remplazadas por caracteres extraños. Si esto ocurre, seleccione la codificación UTF-8. Esto le permitirá resolver el problema, a menos que haya otro tipo de error en los datos.
En la esquina superior derecha, verá un cuadro de texto en el que puede modificar el nombre del proyecto. En ese cuadro, escriba Datos_OR y haga clic en el botón Create Project (Fig. 3). Opcionalmente, puede añadir Tags (etiquetas) para organizar y filtrar los proyectos en la aplicación.
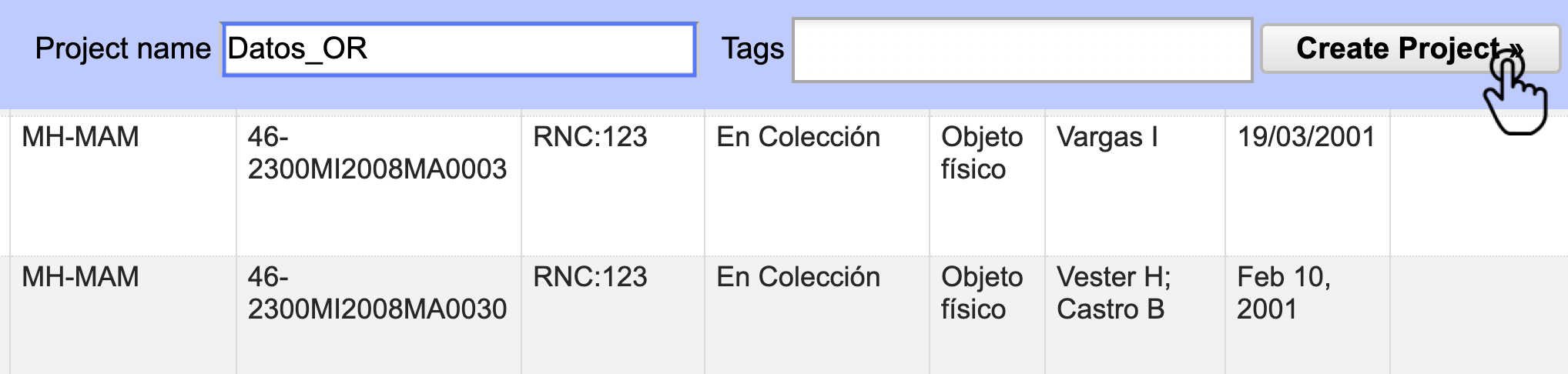
Figura 3. Configuración del proyecto, ajuste de nombre, adición de etiquetas y creación.
Espere a que cargue el archivo. Este proceso puede tomar un tiempo, dependiendo del tamaño que tenga archivo.
Es un método para filtrar los datos en conjuntos más pequeños, facilitando la validación y limpieza de los datos. Puede implementarse para texto, números y fechas.
3.1.1. Crear un Text facet
Diríjase a la columna class, haga clic en el menú de la columna y siga la ruta Facet > Text facet (Fig. 4).
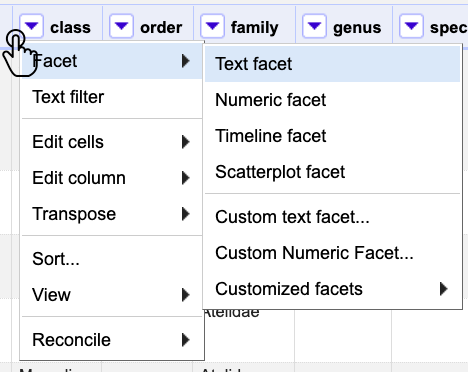
Figura 4. Creación de un Text facet.
3.1.2. Organizar el Text facet
El Text facet creado aparecerá a la izquierda de la aplicación. Verá una ventana con el nombre de la columna y el Facet que acaba de crear (Fig. 5). Haga clic en count para organizar las clases de mayor a menor número de repeticiones y en name para organizarlas en orden alfabético.
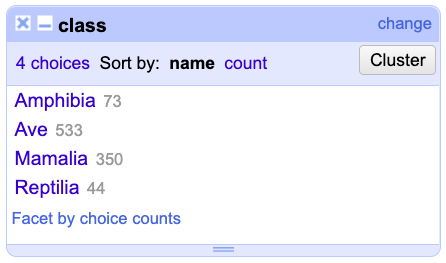
Figura 5. Vista del Text facet y las opciones para organizar las opciones de texto de la columna.
3.1.3. Corrección de los datos
Corrija las inconsistencias en los nombres de las clases Aves y Mammalia. Para esto, acerque el cursor al valor que desea corregir y haga clic en Edit. Luego, en el cuadro de texto que aparece, corrija el error y haga clic en Apply (Fig. 6). Todos los valores serán corregidos de manera automática y las celdas se transformarán de forma masiva.
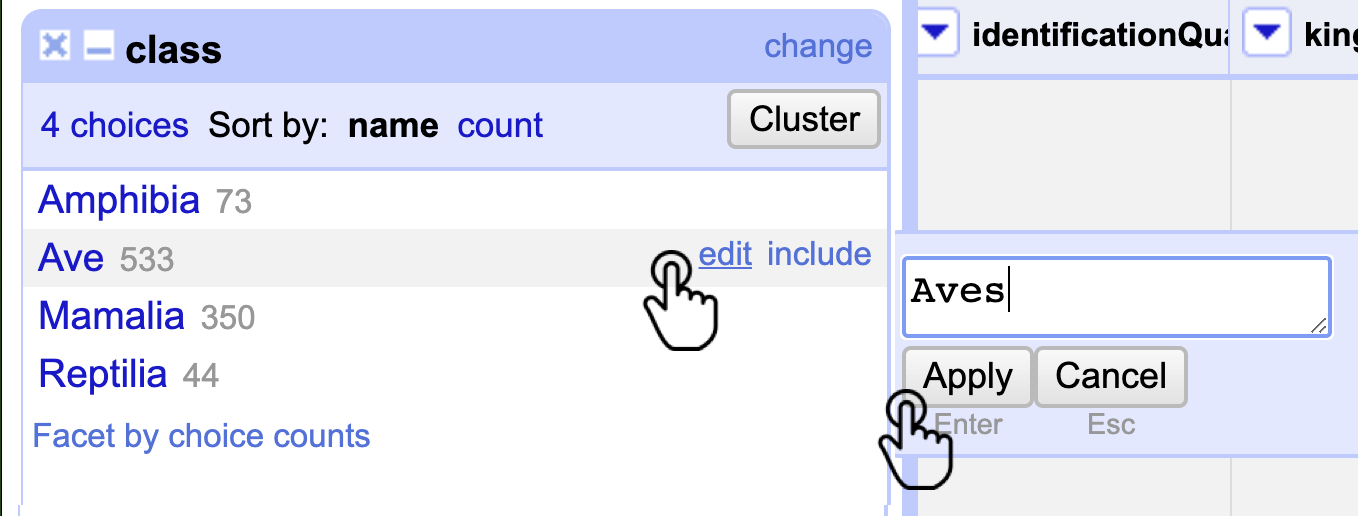
Figura 6. Corrección masiva de la columna class por medio de un Text facet.
Realice el mismo proceso con las columnas basisOfRecord y sex, ajustándolas al vocabulario controlado de cada elemento (Revise el Laboratorio de estandarización) (Fig. 7).
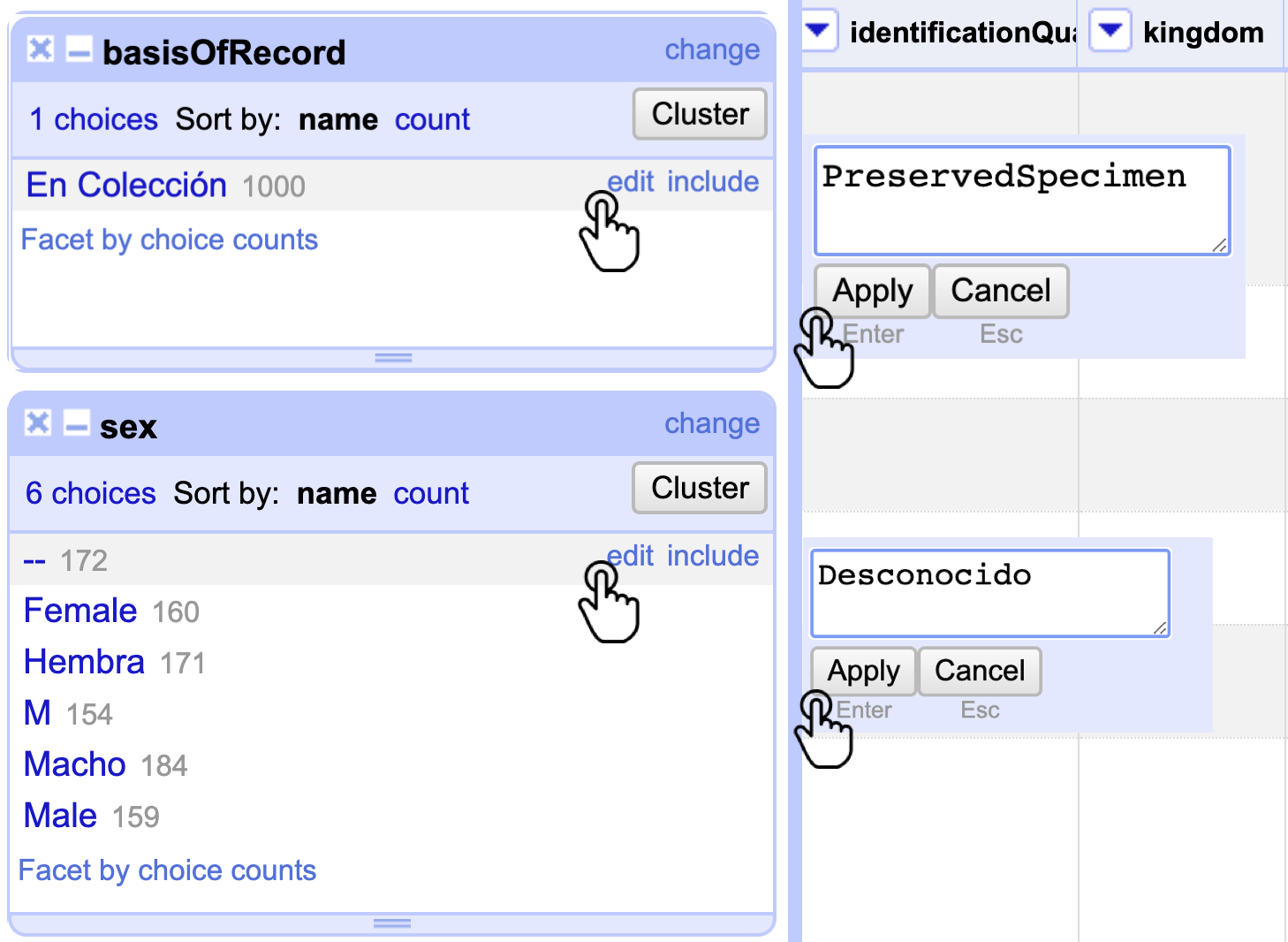
Figura 7. Corrección masiva de las columnas basisOfRecord y sex por medio de un Text facet.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
Figura 8. Remover todos los facets y filtros activos.
Diríjase a la columna individualCount, haga clic en el menú de la columna y realice un Text facet. A su izquierda aparecerá la ventana con el nombre de la columna y el facet que se realizó (Fig. 9).
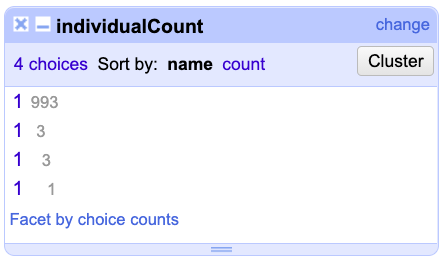
Figura 9. Vista del Text facet y las diferentes formas de documentación del elemento individualCount.
Aunque parece que los datos no tienen errores, este procedimiento evidencia que el programa ha detectado espacios extra. Por tal motivo, el facet muestra cuatro opciones diferentes para el valor “1”. Corrija las inconsistencias desde el menú de la columna individualCount, siguiendo la ruta <span class=”tag is-warning is-light>Edit Cells > Common transforms > Trim leading and trailing whitespace</span>. Posteriormente, verá un mensaje de notificación que indicará en cuántas celdas se eliminaron espacios (Fig. 10).

Figura 10. Transformación de la columna para eliminar dobles espacios y espacios al final del texto.
Si observa la ventana del facet de individualCount, notará que ahora solo existe una opción y los espacios fueron eliminados.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All para quitar todos los facets y filtros que tenga en uso.
Diríjase a la columna catalogNumber, haga clic en el menú de la columna y siga la ruta Facet > Customized facets > Duplicates facet. La ventana del facet aparecerá a la izquierda (Fig. 11).
Se evidencia que el programa ha detectado valores únicos false y valores duplicados true (Fig. 11). Haga clic en true y verá los registros. De esta manera, se pueden detectar los duplicados para un análisis posterior. En este caso, cambie el registro de “Feb 2001” por 46-2300MI2008AV0248, tanto en catalogNumber como en occurrenceIDr (Fig. 11).
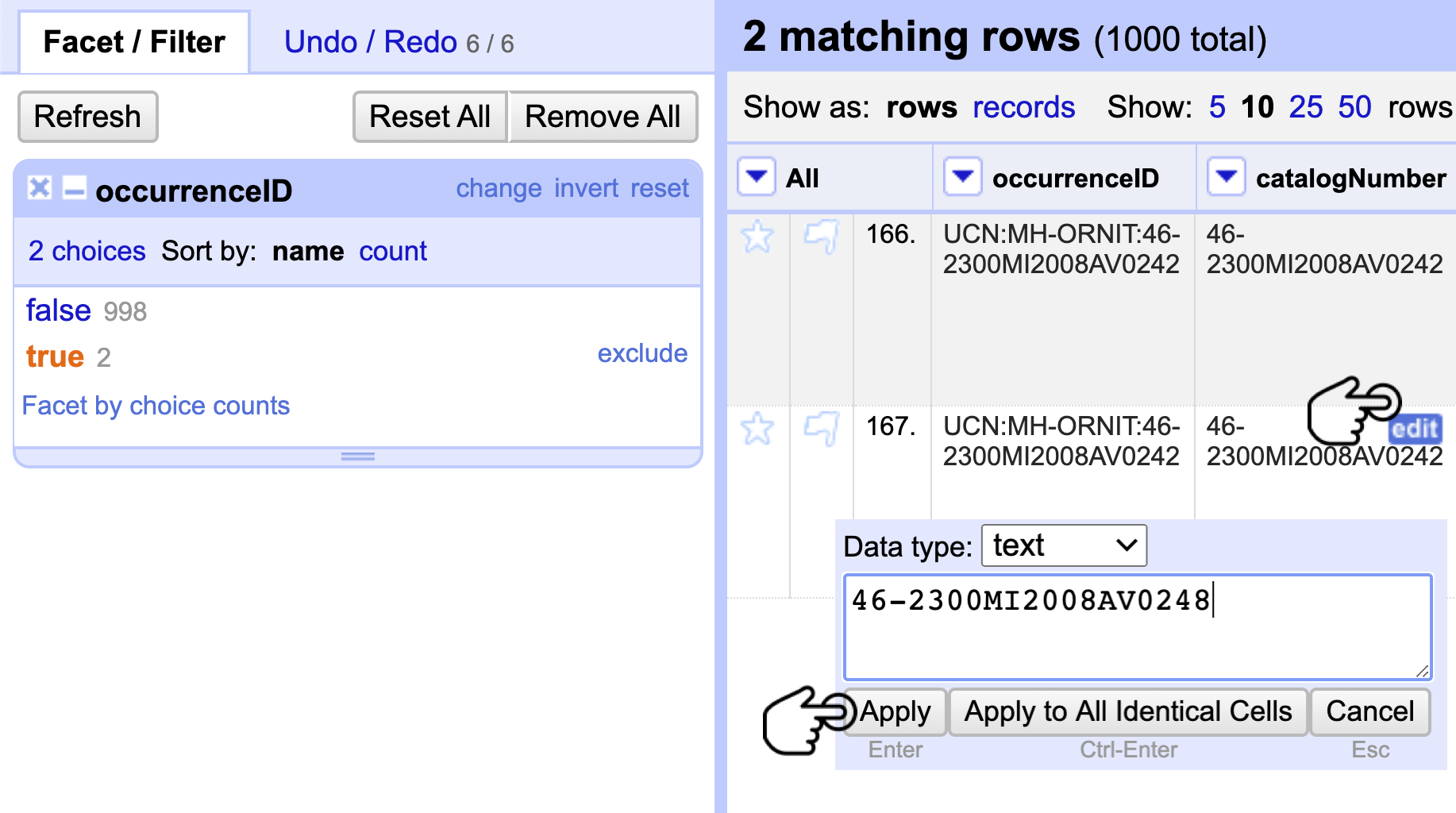
Figura 11. Revisión de los registros duplicados y corrección de los identificadores.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
4.1.1. Corrección elemento scientificName
Diríjase a la columna scientificName, haga clic en el menú de la columna y luego en Text filter. Cuando aparezca la ventana del filtro, escriba sp. en el campo de texto y realice un Text facet en scientificName para visualizar los registros que tienen el valor mencionado (Fig. 12).
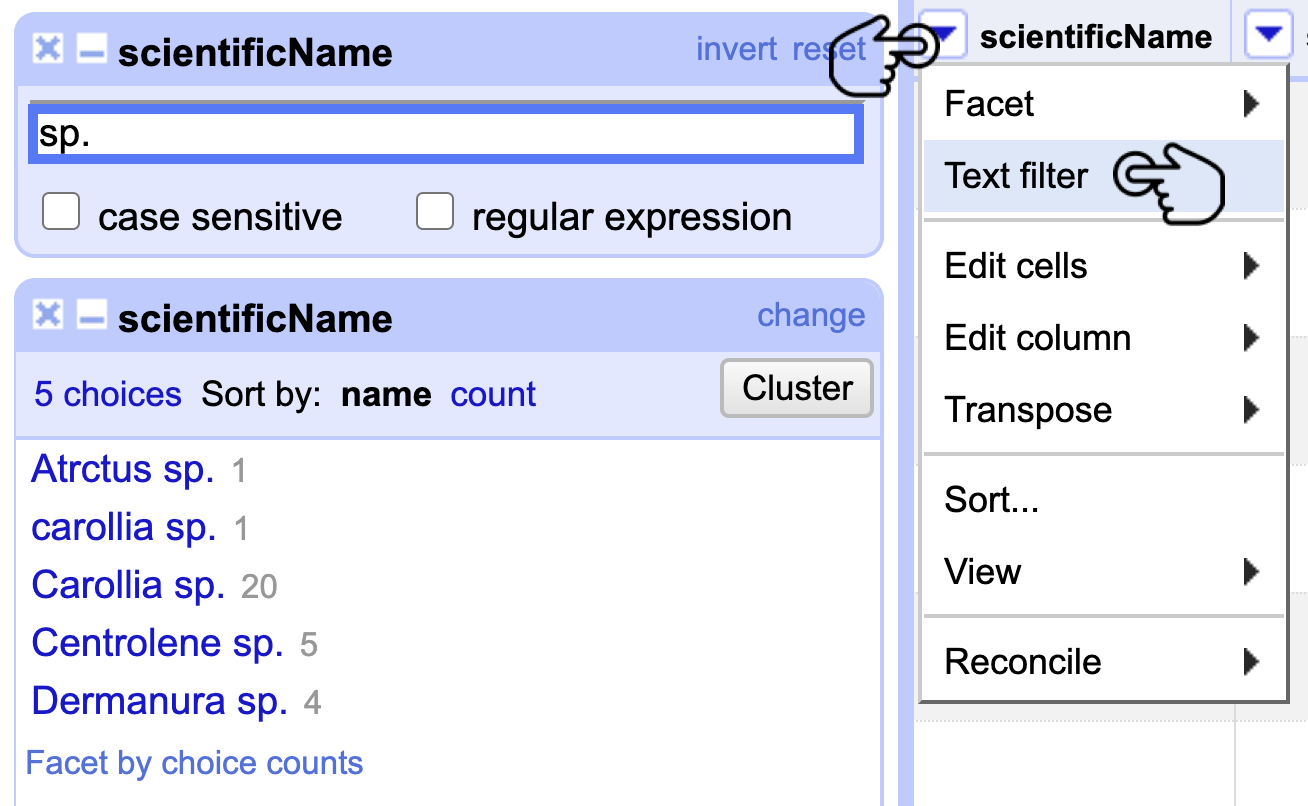
Figura 12. Aplicación de un filtro de texto a la columna scientificName e identificación de nombres científicos a corregir.
Este tipo de identificación parcial (sp.) no debe documentarse en el elemento scientificName, ya que deben registrarse en el elemento DwC verbatimTaxonRank.
Realice un Text facet en verbatimTaxonRank y reemplace masivamente las celdas vacías (blank) con sp.. Después, haga clic en Apply (Fig. 13).
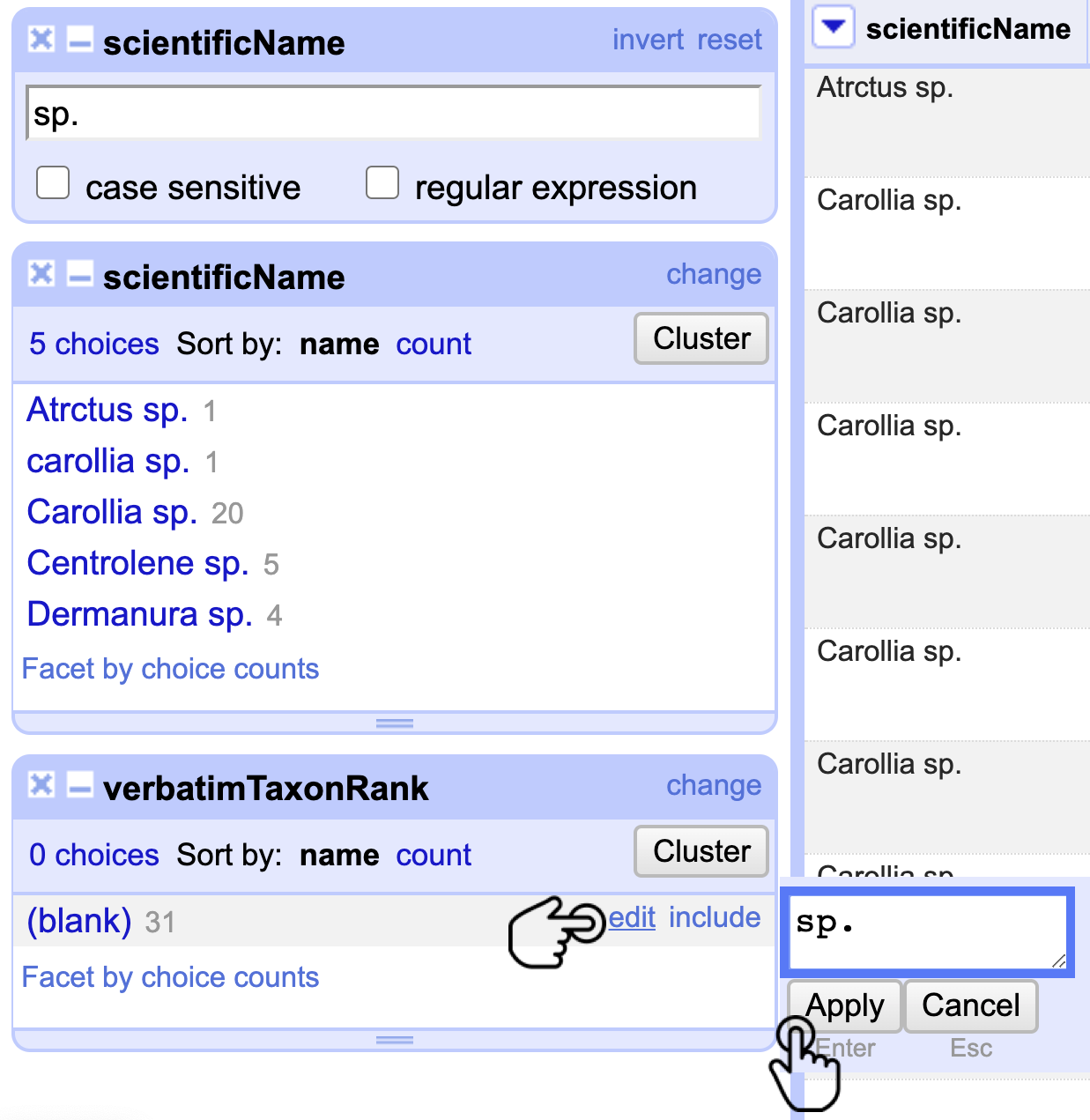
Figura 13. Documentación masiva del elemento verbatimTaxonRank.
Diríjase nuevamente al menú de la columna de scientificName y siga la ruta Edit cells > Transform. Posteriormente, ingrese el comando value.replace en el cuadro de texto “Expression“. Note que la pestaña “Preview“ muestra una vista previa del resultado al aplicar el comando. Corrobore si es el cambio deseado (Fig. 14) y haga clic en OK. De inmediato, aparecerá un mensaje de confirmación de los cambios.
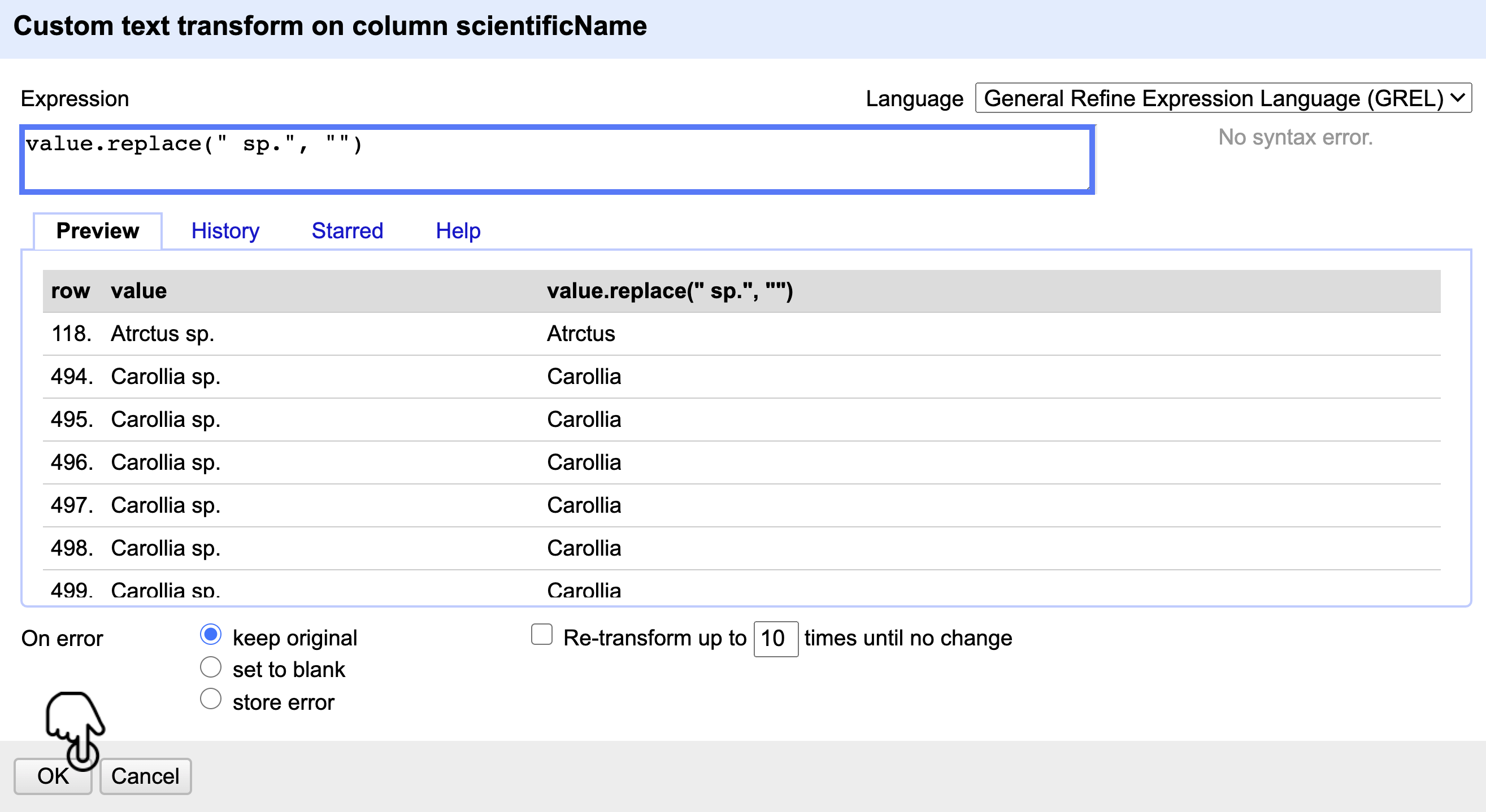
Figura 14. Transformación masiva de los datos con el comando value.replace del lenguaje GREL de OpenRefine.
Utilizando el comando value.replace(“ sp.”,””), se puede sustituir cualquier valor de una columna. Para ello, es necesario cambiar el contenido del paréntesis, escribiendo el valor a buscar encerrado entre comillas “” (ej. “ sp.”) y separado por una coma , . Justo después, se coloca valor de reemplazo entre comillas. En este caso, no hay valores de reemplazo, así que solamente se ponen las comillas “” .
Para conocer más sobre el lenguaje de programación GREL de OpenRefine, diríjase a la documentación de OpenRefine.
4.1.2. Corrección elementos recordedBy e ridentifiedBy
Corrija las columnas recordedBy e ridentifiedBy, empleando el comando value.replace del punto anterior. Para ello, reemplace en ambos elementos el carácter de separación entre los nombres ”;” por el que acepta el estándar Darwin Core para este elemento ” | “. (Para conocer el contexto de este paso, revise el Laboratorio de estandarización).
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
4.2.1. Corrección elemento family
Diríjase a la columna family y realice un Text facet. Haga clic en el menú de la columna y luego en Text filter para que aparezca la ventana del filtro. Marque la casilla regular expression y escriba en el cuadro de texto la expresión .*(?:(?!ae).).$. Esta expresión permite excluir todas las palabras de la columna que no terminan en ae, las cuales corresponden a las últimas letras de la declinación en latín para la categoría taxonómica de familia (idae, ceae) (Fig, 15).
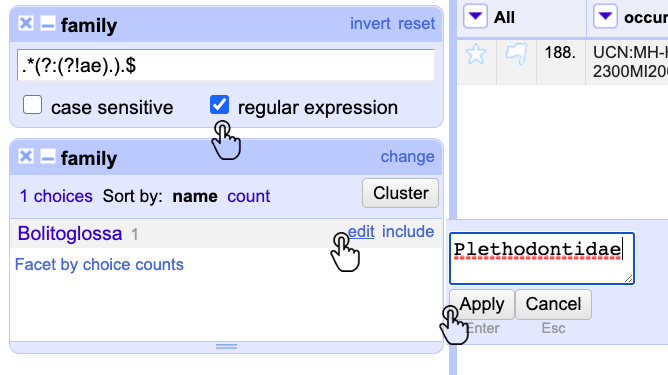
Figura 15. Uso de expresiones regulares en GREL para filtrar la columna family.
Luego de realizar el procedimiento anterior, observará que los registros que no corresponden a la categoría de familia han sido filtrados. Para editarlos, utilice las opciones aprendidas en pasos previos. En este caso particular, reemplace Bolitoglossa, que corresponde a un género, por Plethodontidae, la familia a la que corresponde el nombre científico (Fig. 15).
Para conocer más acerca de las expresiones regulares, visite la documentación de OpenRefine.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
4.2.2. Corrección elementos scientificName e identificationQualifier
Diríjase a la columna scientificName, haga clic en el menú y elija la opción Text filter. Cuando aparezca la ventana del filtro, marque la casilla regular expression y escriba en el campo de texto la expresión [.]. Luego, realice un Text facet para visualizar los registros con este elemento (Fig. 16).
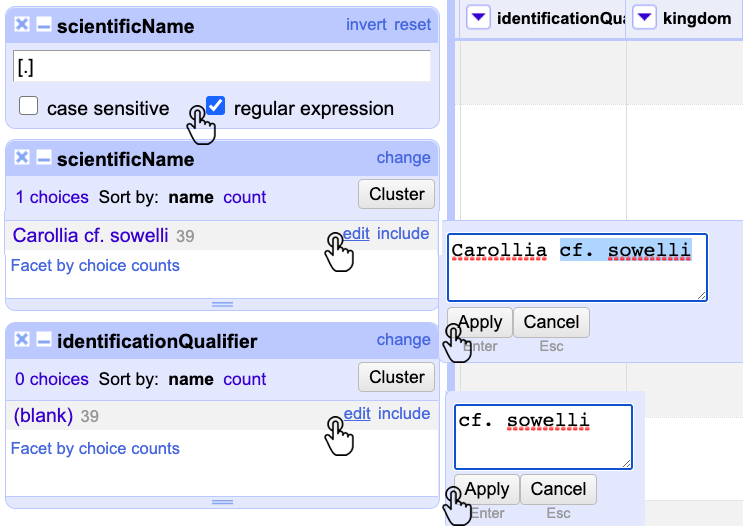
Figura 16. Uso de expresiones regulares en GREL para filtrar y corregir las columnas scientificName e identificationQualifier.
Al terminar el procedimiento anterior, observará los registros que cumplen con el criterio de la expresión regular. Dado que el elemento identificationQualifier está diseñado para almacenar este tipo de información, el elemento scientificName no debe tener estos calificadores.
Para ajustar ambos elementos, aplique un Text facet en identificationQualifier y reemplace el (blank) por cf. sowelli. Además, borre ese mismo texto en el scientificName (Fig. 16). De esta manera, se corregirán masivamente todos los campos.
Finalmente, debe documentarse el género Carollia en scientificName y asociarlo con el calificativo cf. sowelli en identificationQualifier (Fig. 17).
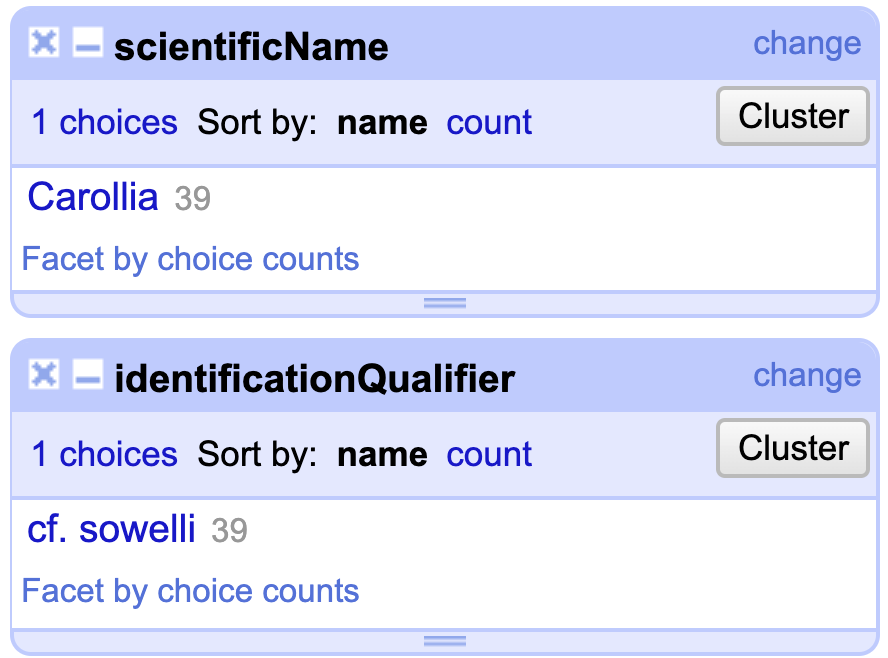
Figura 17. Documentación corregida y correcta de los elementos scientificName e identificationQualifier.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
Diríjase a la columna recordedBy y despliegue el menú. Posteriormente, haga clic en Text facet para que la ventana del facet con más de 200 opciones (choices) diferentes (Fig. 18).
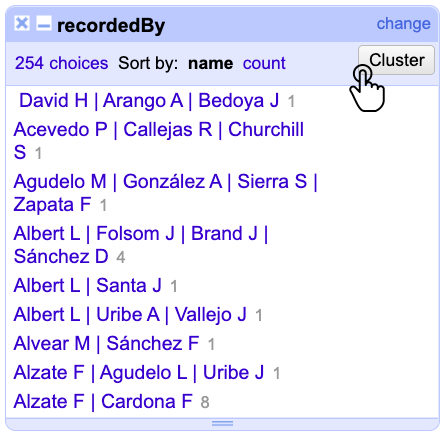
Figura 18. Facet y opciones del elemento recordedBy.
En la parte superior derecha, verá el botón Cluster. Al hacer clic en él (Fig. 18), aparecerá la ventana de “Cluster & Edit“ para la columna recordedBy (Fig. 19).
Podrá ver la siguiente información:
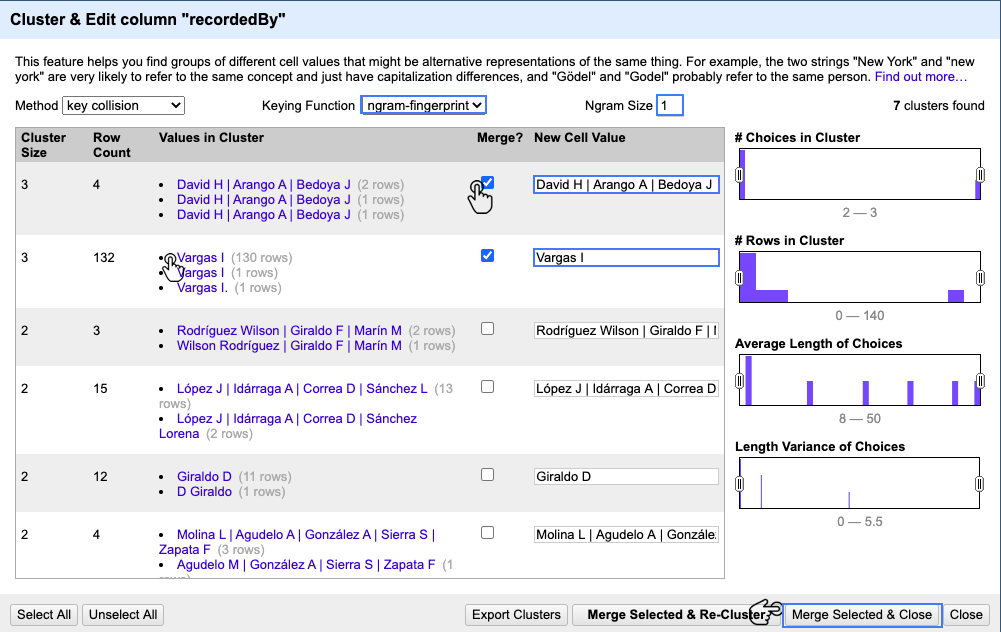
Figura 19. Detalle de la ventana “Cluster & Edit” para hacer realizar clusters y las opciones de configuración disponibles.
Vaya a Keying Function, seleccione ngram-fingerprint y escriba 1 en Ngram Size (Fig. 19).
Para conocer más acerca de los algoritmos, visite la documentación de OpenRefine.
Asigne un valor nuevo para el primer cluster. Para hacerlo, vaya al cuadro de texto de New cell value y escriba David H | Arango A | Bedoya J (con espacios sencillos). Posteriormente, haga check en el cuadro de Merge? para ese cluster (Fig. 19).
Para el segundo cluster, haga clic en Vargas I (la primera opción sin espacios adicionales). Al hacer esto, el valor en New cell value cambiará y la casilla Merge? se marcará automáticamente (Fig. 19).
Con respecto a los clusters restantes, evalúe si se deben agrupar o no, de acuerdo a las opciones disponibles. Si considera que se deben a grupar, marque la casilla Merge?.
Una vez que elija las entradas que desee fusionar en los clusters mencionados, haga clic en Merge Selected & close para agrupar los valores y volver a la ventana principal.
Observe que la cantidad de opciones de datos disminuyó y que la primera entrada de nombres ha cambiado. Es decir, la información se simplificó y organizó correctamente gracias a este proceso.
Al finalizar este ejercicio, diríjase al menú lateral y seleccione la opción Remove All (Fig. 8) para quitar todos los facets y filtros que tenga en uso.
En este ejercicio se utiliza el API (ver definición en el glosario) de GBIF para validar la taxonomía de una lista de nombres.
Árbol taxonómico GBIF
GBIF fácilita un árbol taxonómico robusto a partir de la agrupación de reconciliación de diversos grupos biológicos y proveedores de contenido (Tabla 1), los cuales se encuentran respaldados por comunidades de científicos. Este árbol integra múltiples fuentes de datos, lo que facilita las búsquedas y descargas del portal. Además, se actualiza constantemente.
Tabla 1. Principales fuentes que contribuyen a la consolidación del árbol taxonómico de GBIF.
| Proveedor | Enlace |
|---|---|
| CoL | http://www.catalogueoflife.org/ |
| International Barcode of Life project (iBOL) | https://ibol.org/ |
| World Register of Marine Species (WoRMS) | http://www.marinespecies.org/ |
| Index Fungorum | http://www.indexfungorum.org/ |
| Integrated Taxonomic Information System (ITIS) | http://www.itis.gov/ |
| International Plant Names Index | http://www.ipni.org/ |
| The Paleobiology Database | http://www.paleodb.org/ |
Para acceder a una lista completa de los proveedores y descripción de los mismos, ingrese al GBIF Backbone Taxonomy.
Preparación de los datos
En primer lugar, elimine los facets o filtros que tenga activos. Para tener una aproximación inicial al funcionamiento del API, diríjase a la columna recordedBy y realice un Text facet. Posteriormente, haga clic en la opción count y seleccione al investigador con más registros asociados (Vargas I) (Fig. 20).
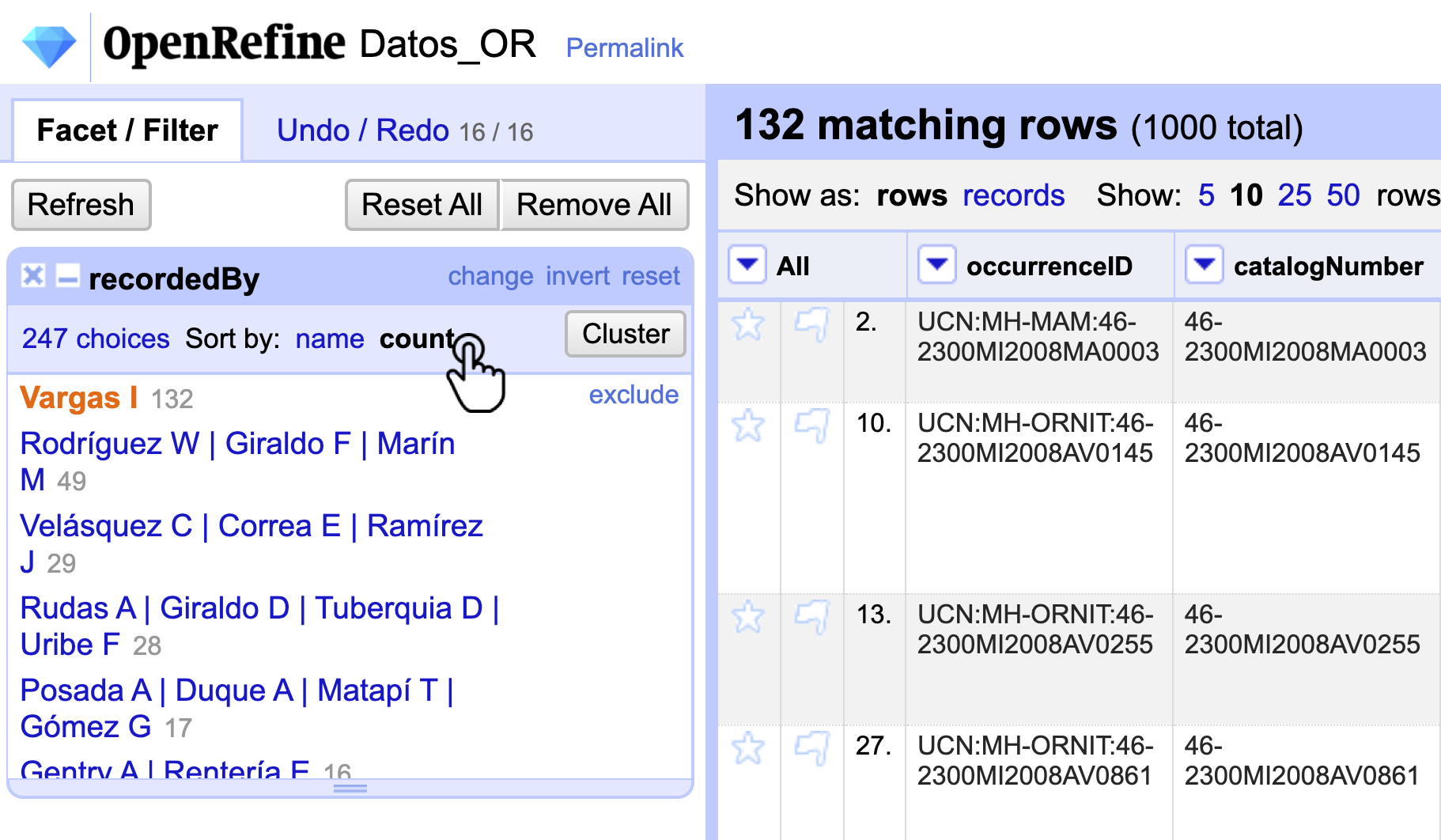
Figura 20. Filtro por conteo en recordedBy y selección de la opción con más registros.
Vaya a la columna scientificName. Es importante que estos nombres no contengan calificadores como “aff.”, “cf.”, “sp.” o “spp.”. Si este el caso, elimínelos como se mostró en los pasos anteriores y deje solamente el nombre científico sin autoría.
Creación de la columna NomAPI
Para realizar la validación a través del API, es necesario que los espacios en blanco de cada nombre científico sean reemplazados por un valor que reconozca el API: %20.
Para ello vaya a Edit column > Add column based on this column y escriba la expresión (tal y como aparece) value.replace(“ “,”%20”). Por último, nombre la columna como “NomAPI” (Fig. 21).
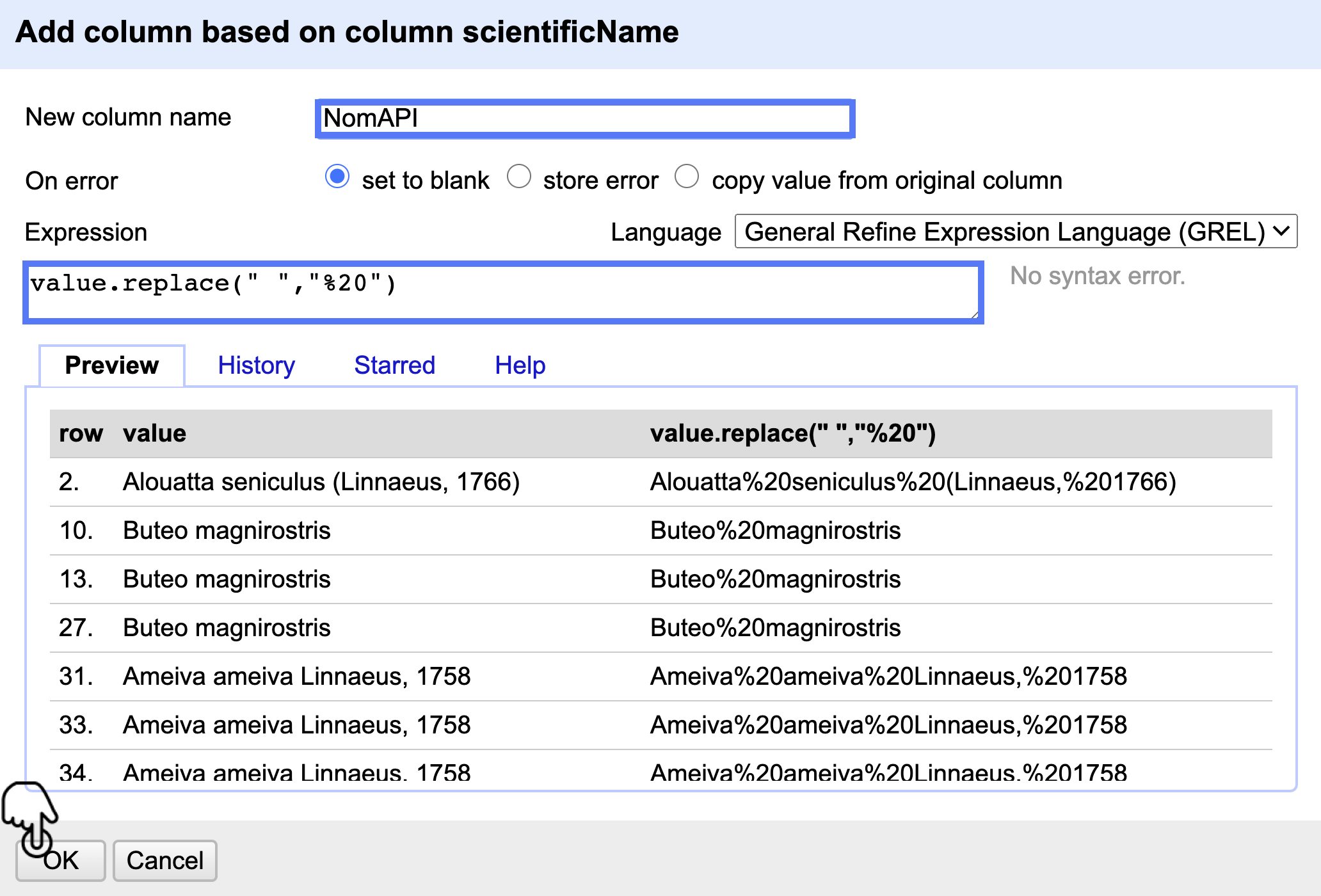
Figura 21. Creación de la nueva columna “NomAPI”.
LLamado al API a partir del nombre científico
Cree una nueva columna llamada “validTax” a partir de la columna “NomAPI”. Para esto, siga la ruta Edit column > Add column by fetching URLs… y escriba la expresión “http://api.gbif.org/v1/species/match?strict=true&name=”+value. Luego,en el cuadro de texto Throttle delay, escriba 5, haga clic en OK y espere a que finalice el proceso (Fig. 22).
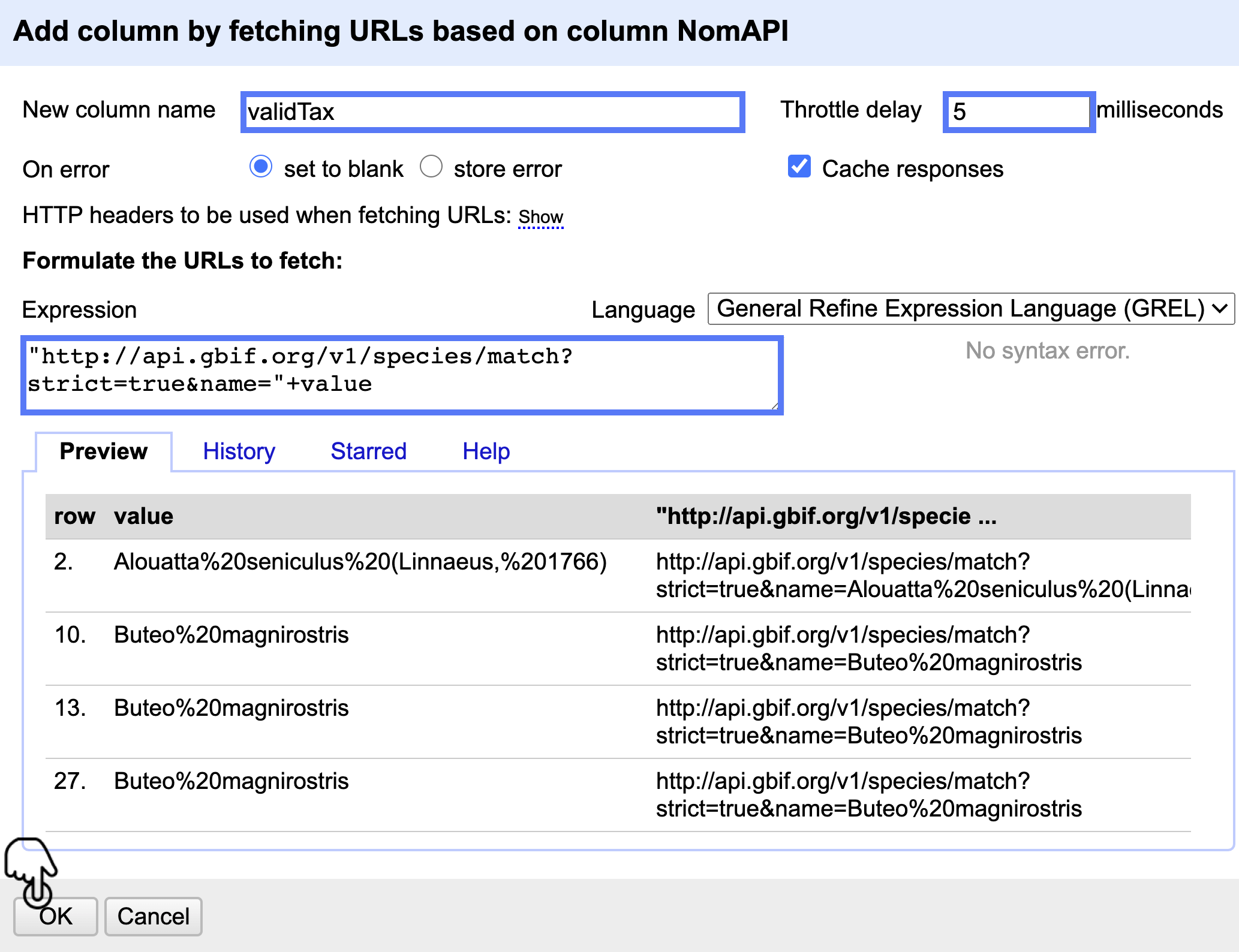
Figura 22. Creación de la nueva columna “validTax”.
Podrá observar que en cada celda de la columna “validTax” aparecen expresiones a partir del llamado al API de GBIF para cada nombre científico consultado (Fig. 23).

Figura 23. Visualización de la columna “validTax”.
Extracción de la información obtenida
Con el fin de evidenciar claramente los resultados de la validación del nombre científico, agregue una columna basada en “validTax”. Para esto, siga la ruta Edit column > Add column based on this column. Luego, nombre la columna como Match y escriba la expresión value.parseJson().get(“matchType”). Por último, haga clic en OK (Fig. 24).
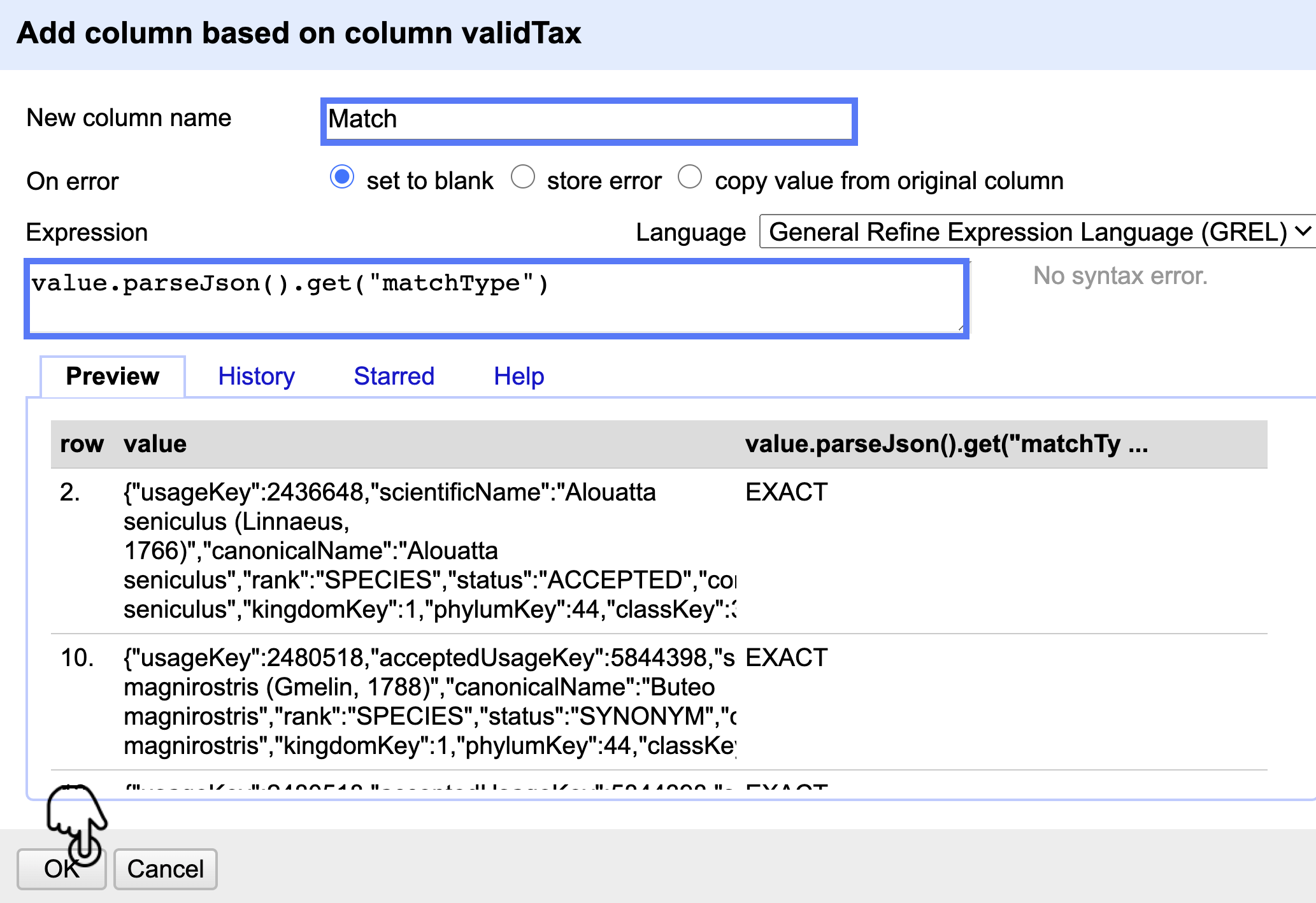
Figura 24. Creación de la columna “Match” a partir de la información obtenida del API.
Validación del nombre científico
Realice un Text facet en la columna “Match” y seleccione Fuzzy. Esta opción denota los nombres científicos con los que no hubo coincidencia exacta entre el conjunto de datos y el árbol taxonómico de GBIF. En este ejercicio, no se encuentra coincidencia total con Dermanura cinereus ni Dermanura glaucus (Fig. 25).
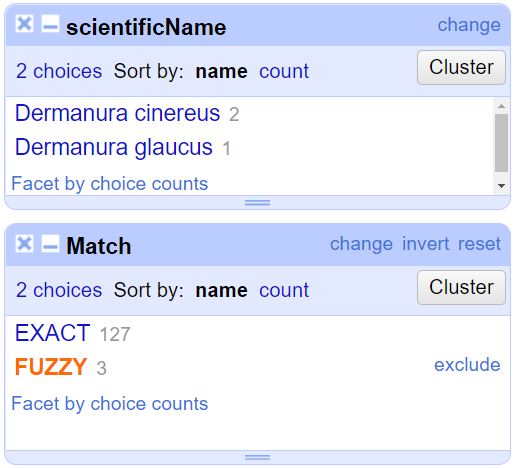
Figura 25. Resultado del facet para Fuzzy.
A través del API, GBIF también ofrece una posible opción de nombres científicos válidos en relación a los que no reconoció totalmente. Es pertinente que evalué estos nombres y decida si deben cambiar.
Agregue una nueva columna llamada “validName” a partir de la columna validTax. Para hacerlo, siga la ruta Edit column > Add column based on this column, escriba la expresión value.parseJson().get(“species”) y haga clic en OK (Fig. 26).
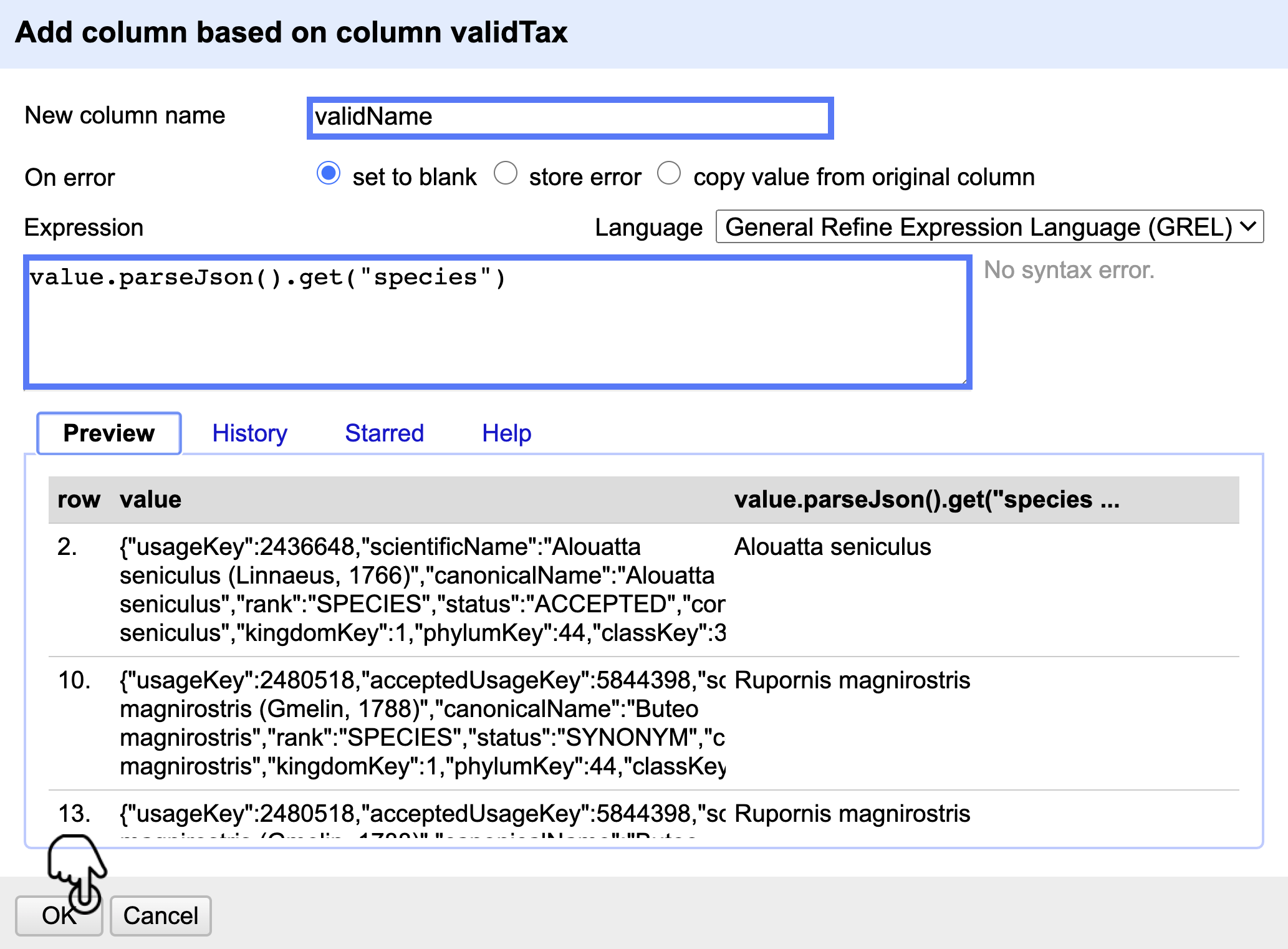
Figura 26. Creación de la nueva columna “validName”.
Haciendo un Text facet en la columna “validName”, verá que GBIF reconoce que las especies a documentar son Artibeus cinereus y Artibereus glaucus, respectivamente (Fig. 27A). Entonces, el paso a seguir es corregir y editar las inconsistencias en la columna scientificName, de acuerdo al “validName” (Fig. 27B).
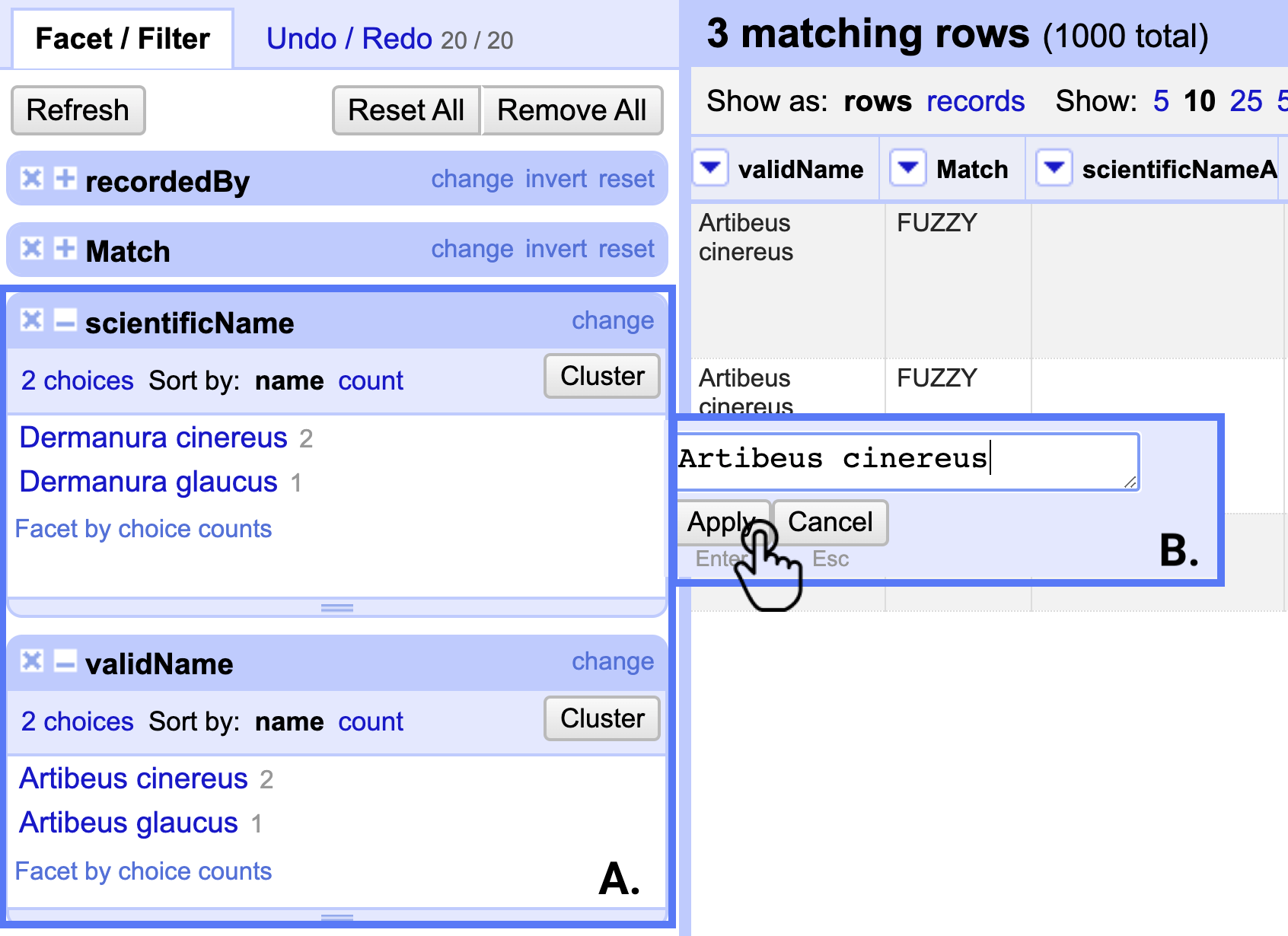
Figura 27. Corrección del nombre científico en OpenRefine. A. Filtro para el nombre científico y la sugerencia de GBIF. B. Corrección en el nombre científico.
Después de realizar el proceso de verificación y la limpieza de nombres científicos, elimine las columnas adicionales que se crearon para este fín (“NomAPI”, “validTax”, “Match” y “validName”). Para ello, siga la ruta Edit column > Remove this column (Fig. 28).
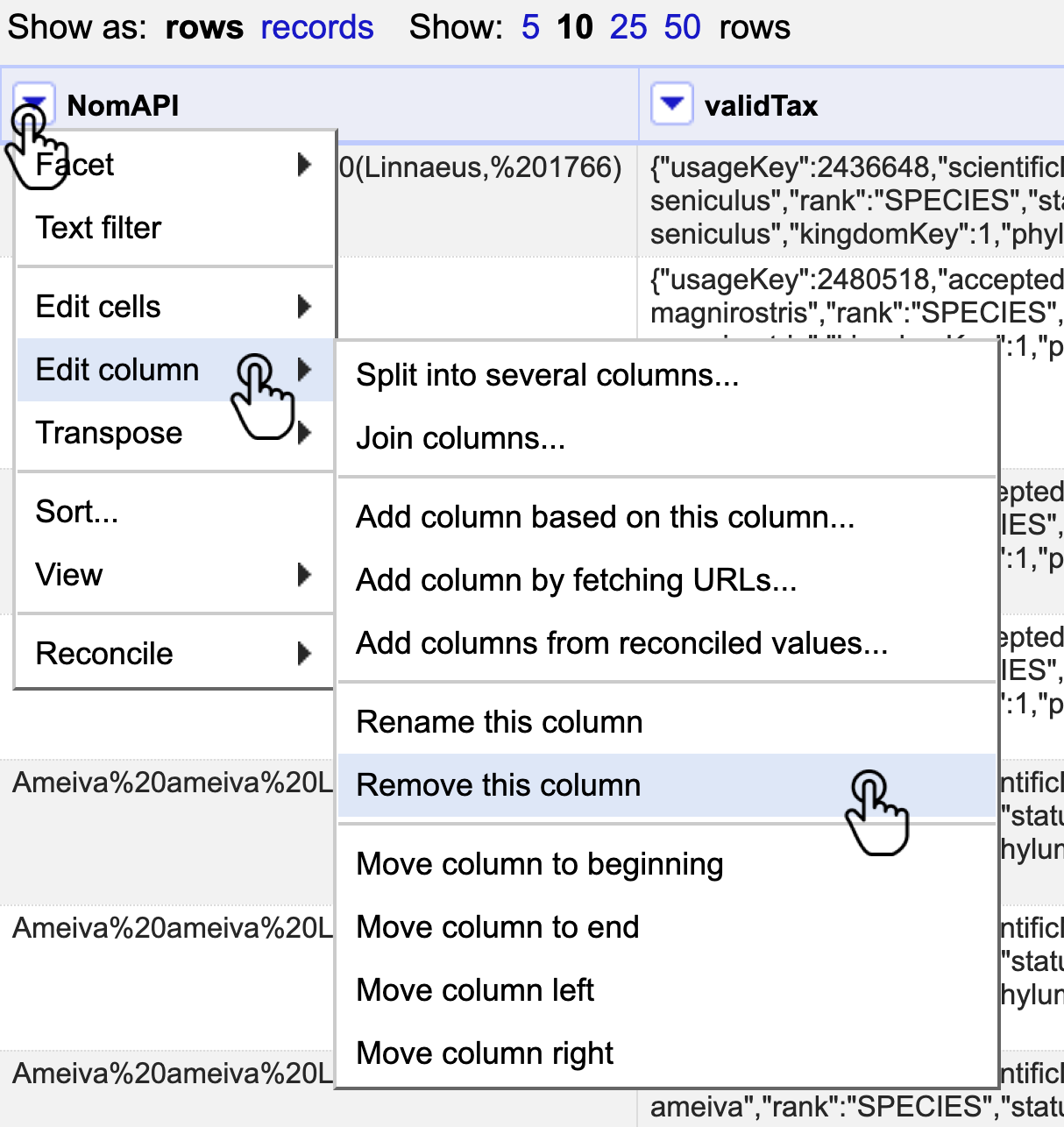
Figura 28. Ruta para eliminar una columna.
Para hacer una modificación en todas las celdas del archivo, diríjase a la primera columna “All”, despliegue el menú y seleccione la opción Transform (Fig. 29).
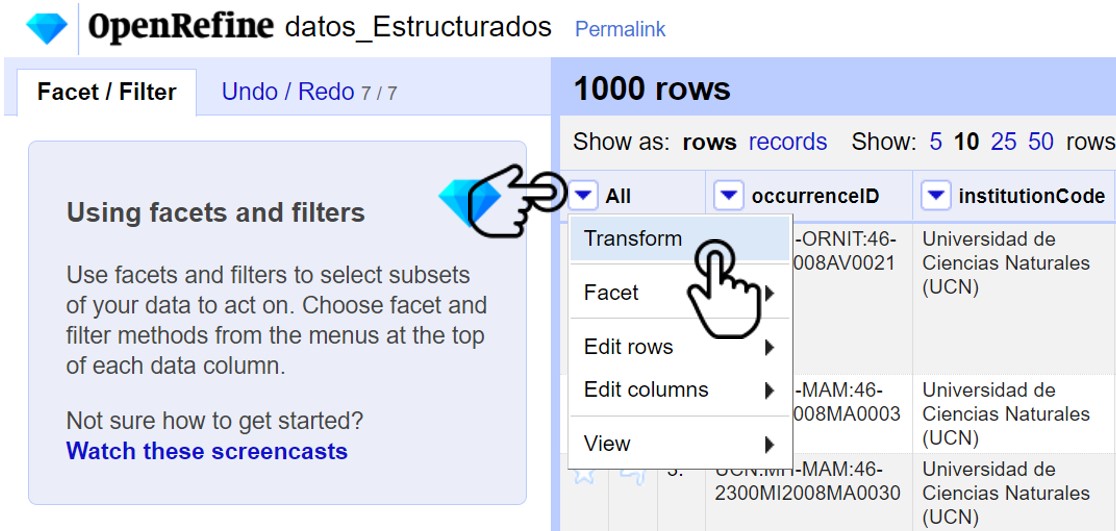
Figura 29. Selección del menú para hacer el cambio masivo.
En la pestaña emergente, introduzca la fórmula value.trim().replace(/\u00A0/,’ ‘).replace(/\s+/,’ ‘). Este comando elimina dobles espacios, saltos de línea y algunos caracteres no reconocibles. Al hacer clic en OK, verá otra pestaña para elegir las columnas a modificar. Deje la opción por defecto con todas las columnas y dé clic en OK (Fig. 30).
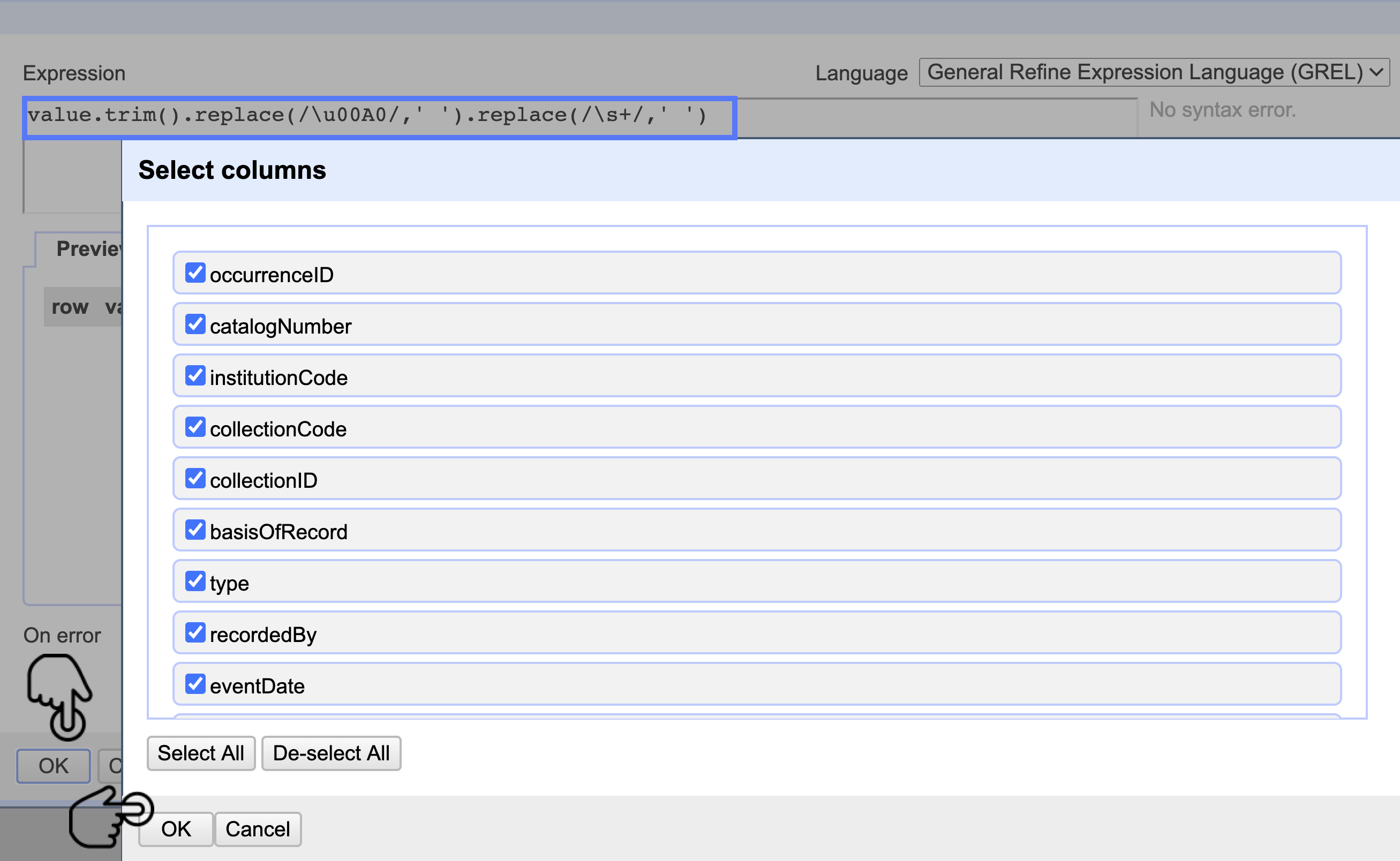
Figura 30. Pestaña para introducir la función de transformación y seleccionar las columnas a las cuales se aplica la función.
Existen múltiples maneras de exportar los archivos en OpenRefine. La siguiente es la más confiable porque cuenta con la mayor cantidad de opciones:
Diríjase a la esquina superior derecha y siga la ruta Export > Custom tabular exporter para acceder a la ventana de exportación (Fig. 31).
En la pestaña “Content” de la ventana emergente, encontrará varias opciones para personalizar la exportación. Algunas de esas opciones son:
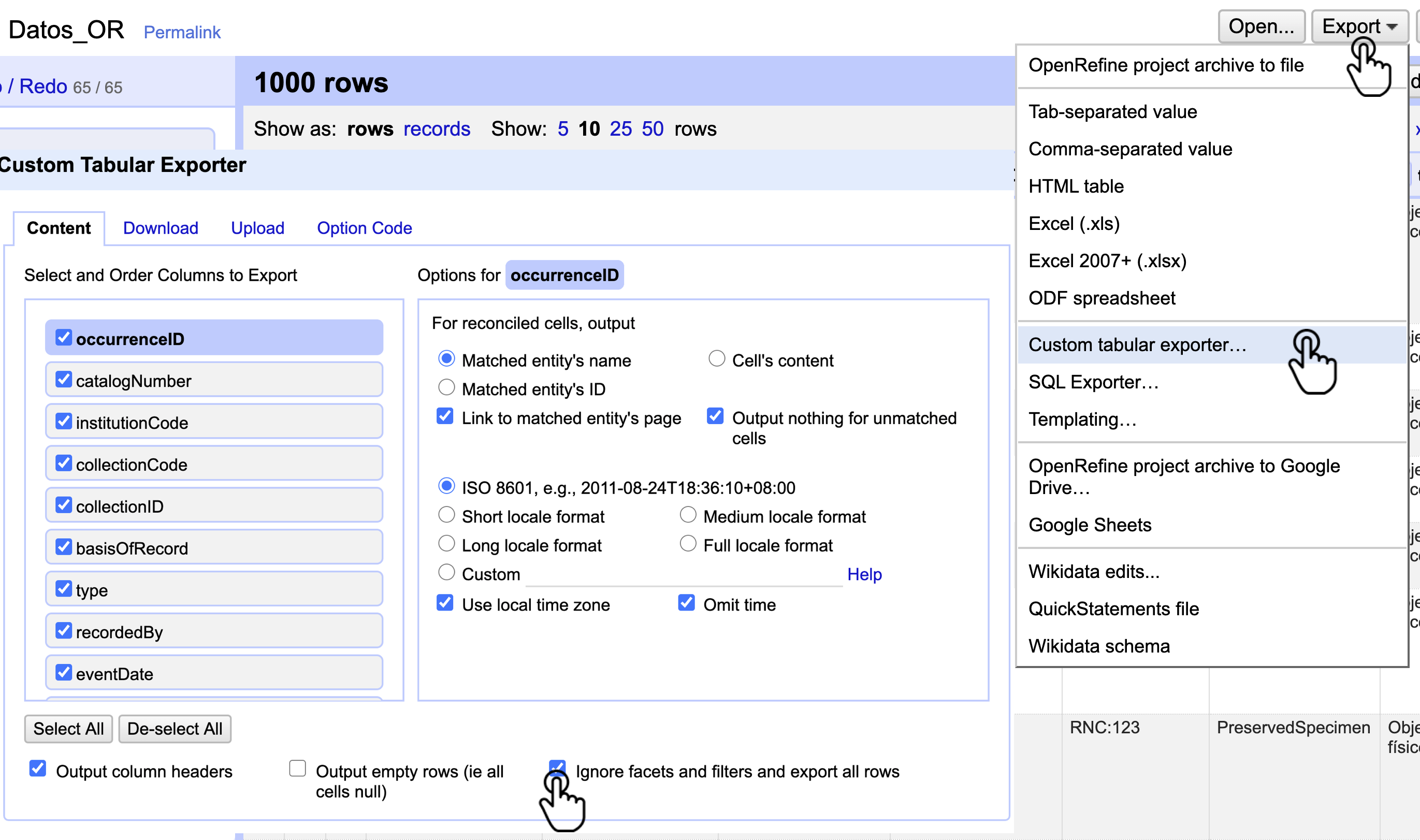
Figura 31. Ruta para expotar el archivo y ventana “content”.
Vaya a la pestaña Download, allí podrá configurar el formano en el cuál se descargan los datos:
Para este caso, ubique el menú “Other formats” y elija la opción Excel in XML(.xlsx). Por último, haga clic en Download y guarde su archivo (Fig. 32).
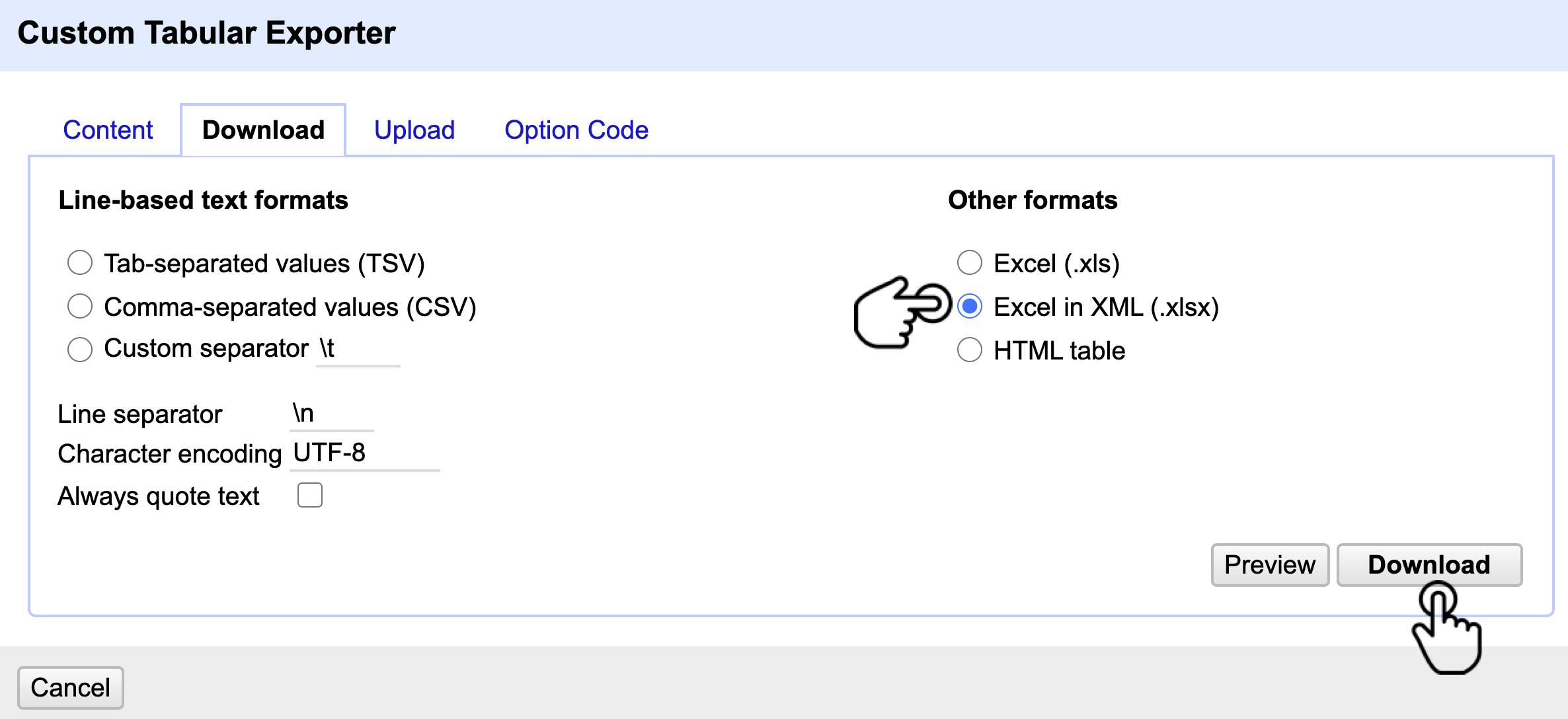
Figura 32. Selección del formato de descarga del conjunto de datos.
OpenRefine también le ofrece la posibilidad de exportar el proyeco completo. Lo anterior permite descargar un archivo que contiene toda la información del proyecto (datos e histórico de cambios). Este archivo se puede utilizar para trabajar en el mismo proyecto de OpenRefine desde otro equipo. Para hacerlo, diríjase a la esquina superior derecha, siga la ruta Export > OpenRefine project archive to file y guarde su archivo (Fig. 33).
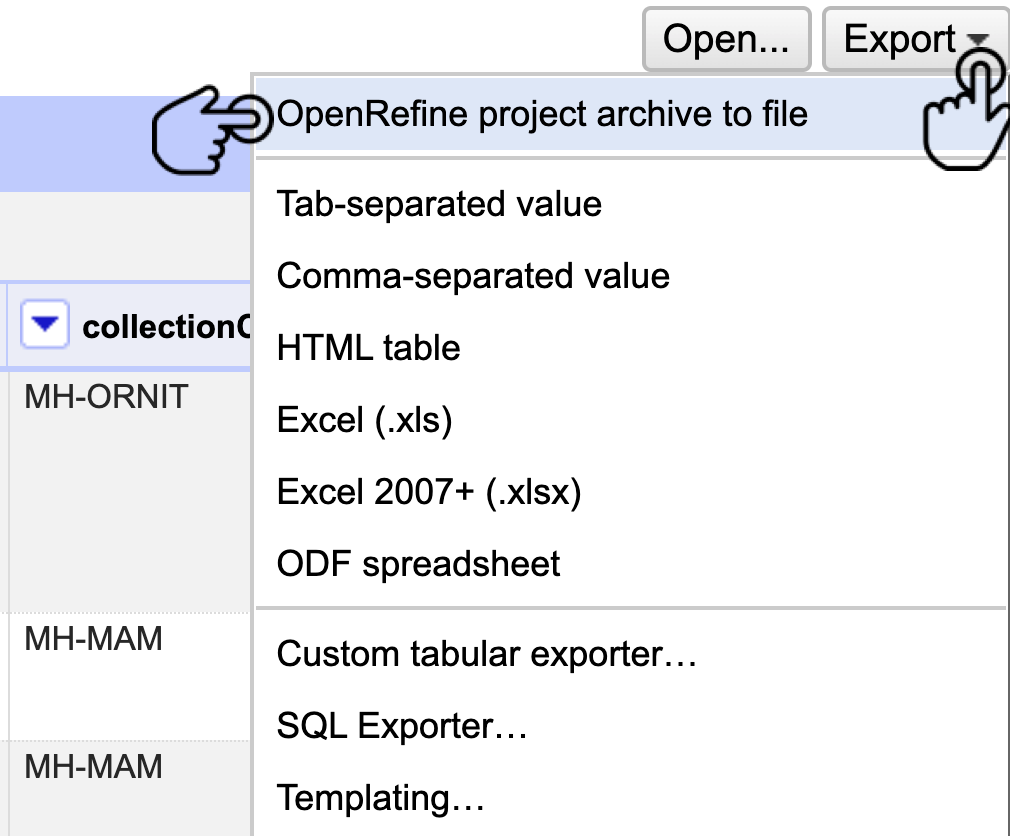
Figura 33. Exportación del proyecto.
Para importar el proyecto, abra OpenRefine y diríjase a la pestaña Import Project. Posteriormente, haga clic en Choose File, seleccione el archivo «Datos_OR.openrefine.tar.gz» y elija la opción Import Project (Fig. 34).
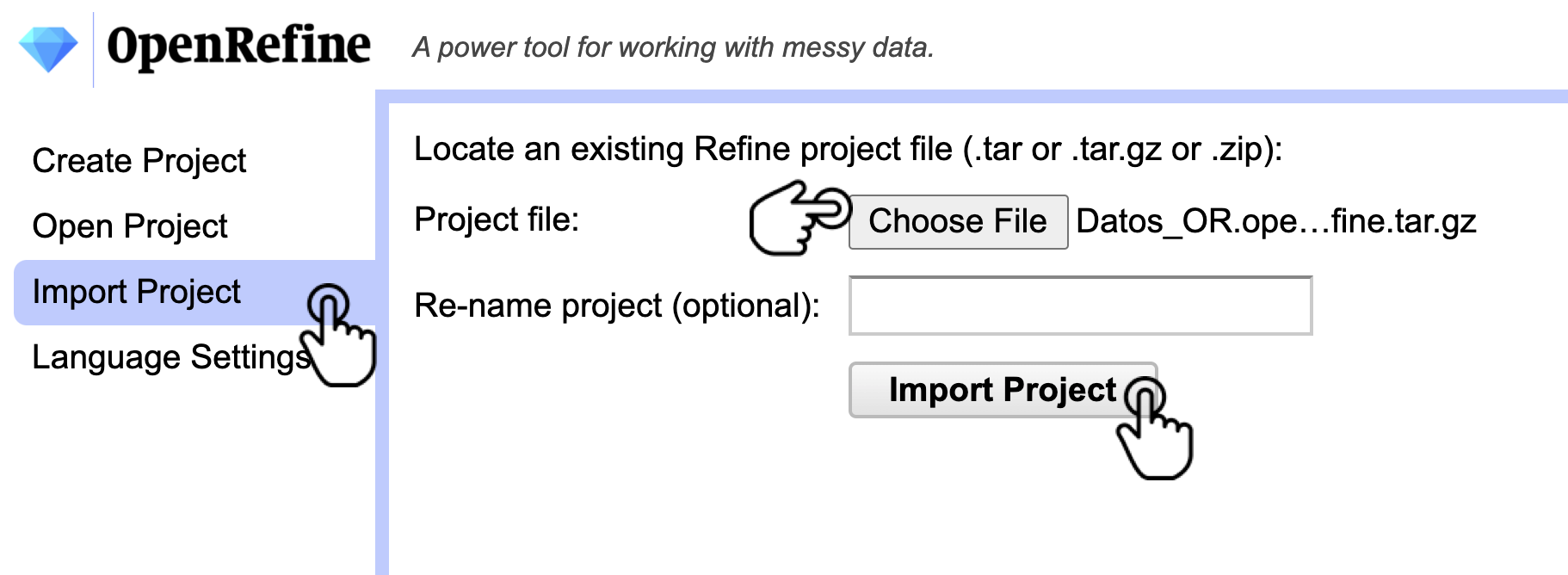
Figura 34. Importación de un proyecto en OpenRefine.
Compare sus resultados con el siguiente archivo, validado según las definiciones del estándar Darwin Core, para identificar aciertos y oportunidades de mejora. Recuerde importarlo como un proyecto en OpenRefine. (Paso 8.2).
¿Qué diferencias encontró con sus resultados?
¡Felicitaciones! Ha aprendido a utilizar OpenRefine para validar y limpiar sus datos sobre biodiversidad.
Atribución y uso de los laboratorios

La licencia CC-BY te permite usar, redistribuir y construir sobre estos contenidos libremente.
¡La difusión de estos laboratorios contribuirá a la publicación de más y mejores conjuntos de datos sobre biodiversidad!
Citación sugerida
Plata C., Ortíz R., Marentes E., Lozano J. (2021). Laboratorio de datos, Ciclo de formación. Consultado a través del SiB Colombia. Disponible en https://biodiversidad.co/formacion/laboratorios.
Fuentes:
Verborgh, R., & De Wilde, M. (2013). Using OpenRefine. Packt Publishing Ltd.
Este sitio web usa cookies, algunas son técnicamente necesarias otras mejoran la experiencia de usuario. Puede rechazar las cookies no esenciales seleccionando “Rechazar”. Consulte la Política de privacidad del sitio web para obtener más información.